Normalisation spatiale d’images de racines de plantes
Encadrants : David Legland, Philippe Andrey
Email : david.legland@grignon.inra.fr
Disponible : OUI
Nombre d'étudiants : 1
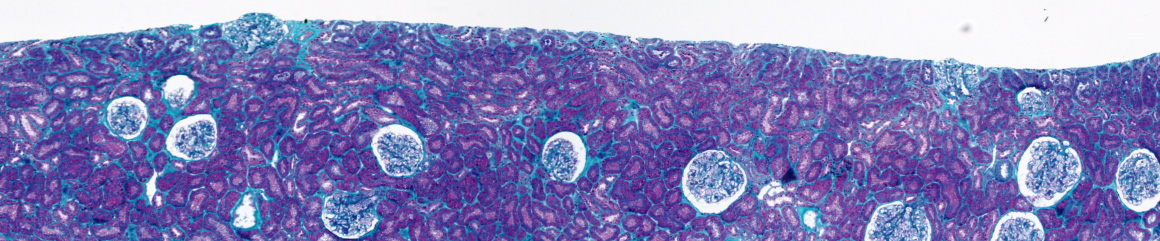
Description : Nous disposons d'une collection d'images 2D obtenues par microscopie confocale dans des racines de plantes. Ces images montrent l'organisation spatiale des cellules dans des domaines racinaires spécifiques. Nous cherchons à mettre en évidence des différences d'organisation entre deux conditions expérimentales.
L’analyse d’images permet de segmenter les objets d’intérêt (les cellules) et de caractériser leur morphologie (forme, taille…). La comparaison de l’organisation cellulaire entre différentes images reste cependant difficile, du fait de la variabilité morphologique des domaines et des cellules. Nous proposons d'aborder cette comparaison par une approche de cartographie statistique, reposant sur une étape de normalisation spatiale rendant possible l’intégration et la fusion des données issues des différentes images
Pré-requis : Les algorithmes seront développés en Java (logiciel ImageJ/Fiji), éventuellement Matlab ou C++, selon le profil de l'étudiant(e).
Travail demandé : Le but du stage est d’implémenter les différentes étapes de cette démarche : cartographie de la morphométrie des cellules, détourage semi-automatique des ensembles de cellules d'intérêt, normalisation spatiale, et enfin fusion des informations extraites des différentes images. Le résultat consistera en des cartes statistiques 2D permettant de comparer différents groupes expérimentaux. Le projet pourra donner suite à un stage dans lequel la démarche sera transposée à l’analyse d’images 3D.
Fichiers complémentaires : projet-IMA_NormaRoot